遺伝子発現機構学研究部門(Closed)
遺伝子発現機構学研究部門ホームページ
私たちの遺伝子発現機構学研究部門では、スプライシングが正確に行われる仕組みを明らかにし、難治疾患において、スプライシング異常がどのように関与しているかを研究しています。小さい研究部門ですが、大きな志を抱いて研究に邁進しております。
遺伝子発現機構学研究部門 サイトはこちら
遺伝子発現機構学研究部門 サイトはこちら
研究内容
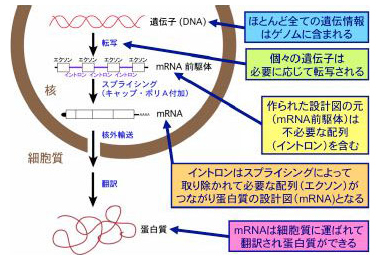
生物の基本単位である細胞の核の中に、生命をつかさどるすべての情報が遺伝子-DNAという物質-に含まれていますが、実際にどのような過程を経て、生物の構造と機能の主役である蛋白質を作っているのでしょうか?
それぞれの細胞において、必要に応じて遺伝子は転写されmRNA前駆体ができ、そこからイントロンと呼ばれている不要な部分が丁寧に取り除かれて、やっと蛋白質の設計図であるmRNAができあがります。mRNAに不必要なイントロンを切り取り、必要なエクソンをつなぎ直す過程を『スプライシング』と呼んでいます(図1)。
それぞれの細胞において、必要に応じて遺伝子は転写されmRNA前駆体ができ、そこからイントロンと呼ばれている不要な部分が丁寧に取り除かれて、やっと蛋白質の設計図であるmRNAができあがります。mRNAに不必要なイントロンを切り取り、必要なエクソンをつなぎ直す過程を『スプライシング』と呼んでいます(図1)。
mRNAの連続した3つの文字(塩基)がアミノ酸に対応するわけですから、スプライシングは正確無比に行われなければ、目的の蛋白質を作れません。ひとたびスプライシングに狂いが生じると、細胞機能に障害をひき起こし、しばしば重い病気の原因になってしまいます。
遺伝子発現機構学研究部門の私たちは、この重要なスプライシングが正確に行われる秘密を明らかにし、スプライシング異常がどのようにして難治疾患を引き起こしているかを研究しています。
遺伝子発現機構学研究部門の私たちは、この重要なスプライシングが正確に行われる秘密を明らかにし、スプライシング異常がどのようにして難治疾患を引き起こしているかを研究しています。
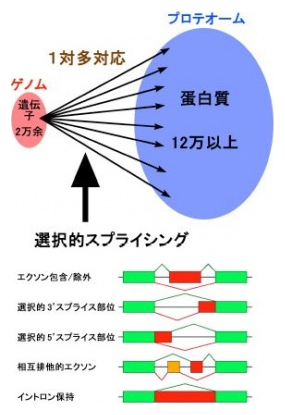
高等生物になるほど長いイントロンが出現し、その数も格段に増えていきます。ヒトでは遺伝子の配列の約99%までがイントロンなので、転写された配列のほとんどを捨て去り、細かくつなぎ合わせた後に、やっと遺伝情報として機能しているのです。イントロンの長さは短いものでは40塩基程度ですが、長いものでは100万塩基を超えています。その長さの違いは実に2万5千倍以上になりますが、スプライシングによって正確に取り除かれているのは紛れもない真実です。スプライシングは当然たいへんな作業を強いられていますが、それは徒労に終わることなく、きわめて重要な意義をもっています。すなわち、1つの遺伝子から転写されたmRNA前駆体は、スプライシングのされ方の違いで複数のmRNAを作ることができるのです。従って、イントロンが多ければ多いほど、スプライシングのされ方の組み合わせは格段に増えていきます。ヒトは2万を少し越えるぐらいの数の遺伝子しか持っていませんが、この『選択的スプライシング』という仕掛けによって、少なくとも12万種の蛋白質が生み出され、高度な生命機能を演出しているのです(左図)。ヒト遺伝子の9割以上が選択的スプライシングされていますが、それはそれぞれの組織や器官の機能に応じて厳格に制御されています。なぜなら、誤った選択的スプライシングをしてしまえば、機能すべき蛋白質ができなくなり、病気の原因となるからです。
私たちが研究している内容は、生命現象の根幹である遺伝子発現の基本的な機能ですが、それが、様々な疾患や癌に密接に関連していることがわかっていただけたと思います。一見地味な基礎研究が、難治疾患の根本原因の解明につながり、それが革新的な根治療法の原理を提供することは歴史が証明しています。私たちは、小さい研究チームですが、大きな志を抱いて日夜研究に邁進しております。
私たちが研究している内容は、生命現象の根幹である遺伝子発現の基本的な機能ですが、それが、様々な疾患や癌に密接に関連していることがわかっていただけたと思います。一見地味な基礎研究が、難治疾患の根本原因の解明につながり、それが革新的な根治療法の原理を提供することは歴史が証明しています。私たちは、小さい研究チームですが、大きな志を抱いて日夜研究に邁進しております。
代表的な業績
K. Fukumura, R. Yoshimoto, L. Sperotto, H-S. Kang, T. Hirose, K. Inoue, M. Sattler & A. Mayeda (2020). SPF45/RBM17-dependent, but not U2AF-dependent, splicing in human short introns. Nat. Commun, in revision (preprint: https://www.biorxiv.org/content/10.1101/784868v3).
R. Yoshimoto, K. Rahimi, T.B. Hansen, J. Kjems & A. Mayeda (2020). Biosynthesis of circular RNA ciRS-7/CDR1as is mediated by Mammalian-wide Interspersed Repeats. iScience 23, 101345.
T. Kameyama, H. Suzuki & A. Mayeda (2012). Re-splicing of mature mRNA in cancer cells promotes activation of distant weak alternative splice sites. Nucleic Acids Res. 40, 7896–7906.
N. Sasaki-Haraguchi, M.K. Shimada, I. Taniguchi, M. Ohno & A. Mayeda (2012). Mechanistic insights into human pre-mRNA splicing of human ultra-short introns: Potential unusual mechanism identifies G-rich introns. Biochem. Biophys. Res. Commun. 423, 289–294.
K. Ohe & A. Mayeda (2010). HMGA1a trapping of U1 snRNP at an authentic 5’ splice site induces aberrant exon skipping in sporadic Alzheimer's disease. Mol. Cell. Biol. 30, 2220–2228.
E. Sakashita, S. Tatsumi, D. Werner, H. Endo & A. Mayeda (2004). Human RNPS1 and its associated factors: a versatile alternative pre-mRNA splicing regulator in vivo. Mol. Cell. Biol. 24, 1174–1187.
T. Manabe, T. Katayama*, N. Sato, F. Gomi, J. Hitomi, T. Yanagida, T. Kudo, A. Honda, Y. Mori, S. Matsuzaki, K. Imaizumi, A. Mayeda* & M. Tohyama (2003). Induced HMGAIa expression causes aberrant splicing of presenilin-2 pre-mRNA in sporadic Alzheimer’s disease. Cell Death Differ. 10, 698–708. (*Corresponding authors)
J. Zhu, A.Mayeda & A.R. Krainer (2001). Exon identity established through differential antogonism between exonic splicing silencer-bound hnRNP A1 and enhancer-bound SR proteins. Mol. Cell 8, 1351–1361.
A. Mayeda*, J. Badolato, R. Kyobayashi, M.Q. Zhang, E.M. Gardiner, & A.R. Krainer (1999). Purification and characterization of human RNPS1: a general activator of pre-mRNA splicing. EMBO J. 18, 4560–4570. (*Corresponding author)
A. Mayeda, G.R. Screaton S.D. Chandler, X-D. Fu & A.R. Krainer (1999). Substrate specificities of SR proteins in constitutive splicing are determined by their RNA-Recognition Motifs and composite pre-mRNA exonic elements. Mol. Cell. Biol. 19, 1853–1863.
A. Mayeda, S.H. Munroe, R.-M. Xu & A.R. Krainer (1998). Distinct functions of the closely related tandem RNA-recognition motifs of hnRNP A1. RNA 4, 1111–1123.
A. Mayeda, S.H. Munroe, J.F. Cáceres & A.R. Krainer (1994). Function of conserved domains of hnRNP A1 and other hnRNP A/B group proteins. EMBO J. 13, 5483–5495.
Q. Sun, A. Mayeda, R.K. Hampson, A.R. Krainer & F.M. Rottman (1993). General splicing factor SF2/ASF promotes alternative splicing by binding to an exonic splicing enhancer. Genes Dev. 7, 2598–2608.
A. Mayeda, D.M. Helfman & A.R. Krainer (1993). Modulation of exon skipping and inclusion by heterogeneous nuclear ribonucleoprotein A1 and pre-mRNA splicing factor SF2. Mol. Cell. Biol. 13, 2993–3001.
A. Mayeda & A.R. Krainer (1992). Regulation of alternative pre-mRNA splicing by hnRNP A1 and splicing factor SF2. Cell 68, 365–375.
A.R. Krainer, A. Mayeda, D. Kozak & G. Binns (1991). Functional Expression of Cloned Human Splicing Factor SF2: Homology to RNA Binding Proteins, U1 70 K, and Drosophila Splicing Regulators. Cell 66, 383–394.
R. Yoshimoto, K. Rahimi, T.B. Hansen, J. Kjems & A. Mayeda (2020). Biosynthesis of circular RNA ciRS-7/CDR1as is mediated by Mammalian-wide Interspersed Repeats. iScience 23, 101345.
T. Kameyama, H. Suzuki & A. Mayeda (2012). Re-splicing of mature mRNA in cancer cells promotes activation of distant weak alternative splice sites. Nucleic Acids Res. 40, 7896–7906.
N. Sasaki-Haraguchi, M.K. Shimada, I. Taniguchi, M. Ohno & A. Mayeda (2012). Mechanistic insights into human pre-mRNA splicing of human ultra-short introns: Potential unusual mechanism identifies G-rich introns. Biochem. Biophys. Res. Commun. 423, 289–294.
K. Ohe & A. Mayeda (2010). HMGA1a trapping of U1 snRNP at an authentic 5’ splice site induces aberrant exon skipping in sporadic Alzheimer's disease. Mol. Cell. Biol. 30, 2220–2228.
E. Sakashita, S. Tatsumi, D. Werner, H. Endo & A. Mayeda (2004). Human RNPS1 and its associated factors: a versatile alternative pre-mRNA splicing regulator in vivo. Mol. Cell. Biol. 24, 1174–1187.
T. Manabe, T. Katayama*, N. Sato, F. Gomi, J. Hitomi, T. Yanagida, T. Kudo, A. Honda, Y. Mori, S. Matsuzaki, K. Imaizumi, A. Mayeda* & M. Tohyama (2003). Induced HMGAIa expression causes aberrant splicing of presenilin-2 pre-mRNA in sporadic Alzheimer’s disease. Cell Death Differ. 10, 698–708. (*Corresponding authors)
J. Zhu, A.Mayeda & A.R. Krainer (2001). Exon identity established through differential antogonism between exonic splicing silencer-bound hnRNP A1 and enhancer-bound SR proteins. Mol. Cell 8, 1351–1361.
A. Mayeda*, J. Badolato, R. Kyobayashi, M.Q. Zhang, E.M. Gardiner, & A.R. Krainer (1999). Purification and characterization of human RNPS1: a general activator of pre-mRNA splicing. EMBO J. 18, 4560–4570. (*Corresponding author)
A. Mayeda, G.R. Screaton S.D. Chandler, X-D. Fu & A.R. Krainer (1999). Substrate specificities of SR proteins in constitutive splicing are determined by their RNA-Recognition Motifs and composite pre-mRNA exonic elements. Mol. Cell. Biol. 19, 1853–1863.
A. Mayeda, S.H. Munroe, R.-M. Xu & A.R. Krainer (1998). Distinct functions of the closely related tandem RNA-recognition motifs of hnRNP A1. RNA 4, 1111–1123.
A. Mayeda, S.H. Munroe, J.F. Cáceres & A.R. Krainer (1994). Function of conserved domains of hnRNP A1 and other hnRNP A/B group proteins. EMBO J. 13, 5483–5495.
Q. Sun, A. Mayeda, R.K. Hampson, A.R. Krainer & F.M. Rottman (1993). General splicing factor SF2/ASF promotes alternative splicing by binding to an exonic splicing enhancer. Genes Dev. 7, 2598–2608.
A. Mayeda, D.M. Helfman & A.R. Krainer (1993). Modulation of exon skipping and inclusion by heterogeneous nuclear ribonucleoprotein A1 and pre-mRNA splicing factor SF2. Mol. Cell. Biol. 13, 2993–3001.
A. Mayeda & A.R. Krainer (1992). Regulation of alternative pre-mRNA splicing by hnRNP A1 and splicing factor SF2. Cell 68, 365–375.
A.R. Krainer, A. Mayeda, D. Kozak & G. Binns (1991). Functional Expression of Cloned Human Splicing Factor SF2: Homology to RNA Binding Proteins, U1 70 K, and Drosophila Splicing Regulators. Cell 66, 383–394.
